PCR을 배우게 되면 가장 먼저 하는게
내가 원하는 바이러스에 대한 primer를 디자인하는 것이다.
손으로 직접 하는 방법도 있지만 프로그램을 이용하면
훨씬 간단하게 알아낼 수 있다.
오늘은 그 방법을 소개하고자 한다.
1.
먼저, target 바이러스를 NCBI에 들어가서 검색해준다.
National Center for Biotechnology Information
www.ncbi.nlm.nih.gov
검색창에 내가 원하는 바이러스 이름+complete genome으로 검색해준다.
EX)
예를 들어 porcine parvovirus 바이러스에 대해 pcr을 하자
그러면 검색에 porcine parvovirus complete genome을 검색한다.
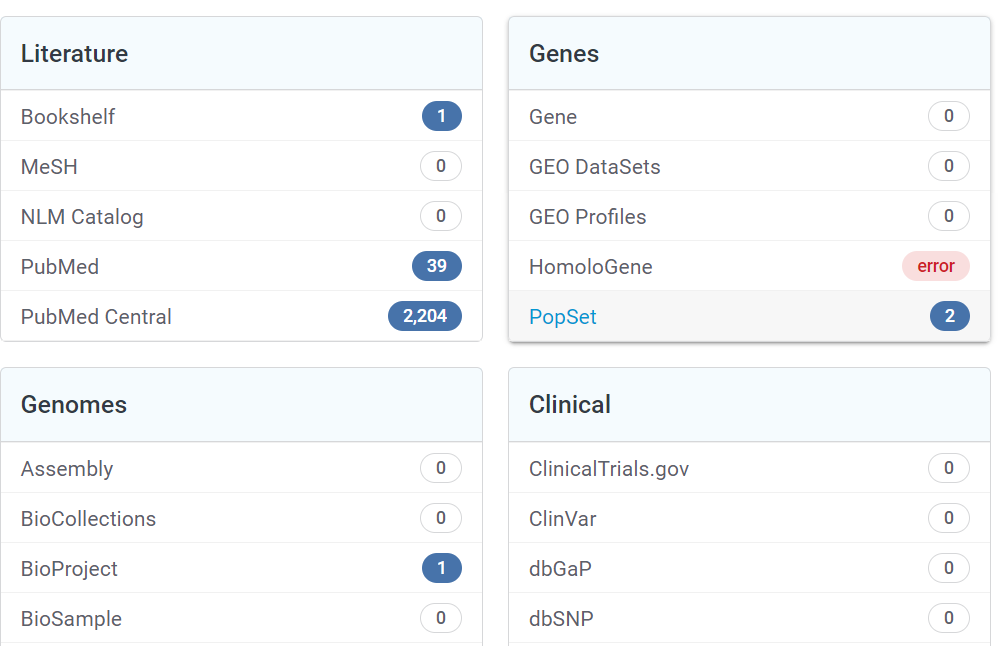
이런 화면을 찾을 수 있는데 여기서 Genomes - Nucleotide에 들어가서
Porcine parvovirus, complete genome 이라 적힌 (isolate X) 6번을 클릭해보면
원하는 바이러스의 origin 전체를 볼 수 있다.
2.

이렇게 바이러스의 정보를 확인할 수 있는데,
바이러스 이름 및에 적힌 NCBI number도 확인할 수 있다.
NCBI reference sequence: NC_001718.1
3.
다음으로 primer를 디자인 할 건데,
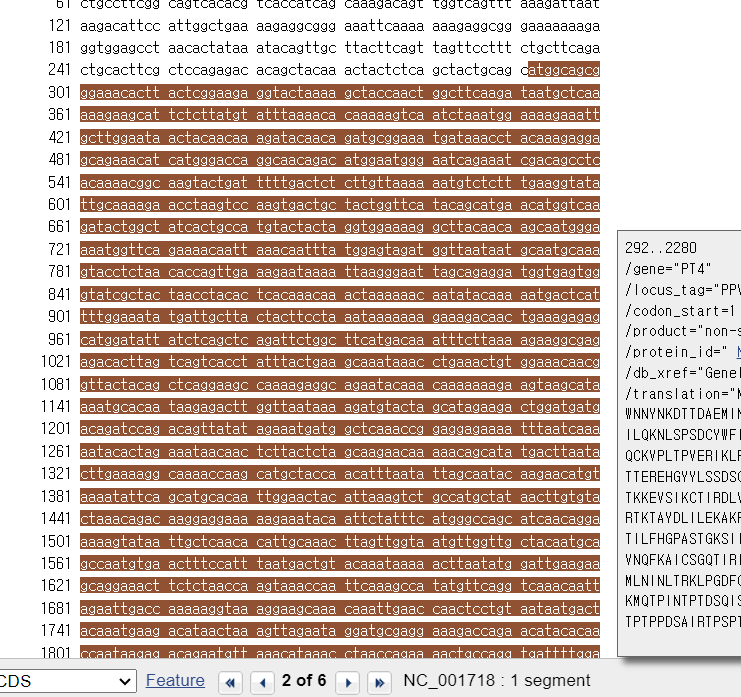
CDS를 선정할 때, /product가 non-structure P인 것을 선택한다.
non-structure가 변이가 적어서 선정한다고 한다.
원하는 cds를 선정하고 클릭하면 아래 서열로 이동된다.
4.
구글에 primer 3을 검색하면
primer3 input이라는 사이트가 나온다.
https://bioinfo.ut.ee/primer3-0.4.0/
Primer3 Input (version 0.4.0)
bioinfo.ut.ee
사이트에 들어가서 갈색부위가 온전한 부위
사진에서는 301번부터 복사해서 붙여넣는다.

가운데 있는 pick hydridization probe도 켜준다.
사진 가장 아래 있는 product size ranges를 150-250으로 수정해준다.
(서열이 5줄 이상이어야 한다.)
다음으로 pick primer 버튼을 눌러준다.
5.

그러면 이런 화면으로 넘어오게 된다.
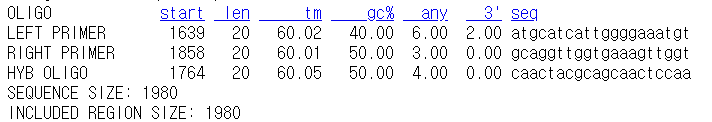
Left primer = forward primer
Right primer = reverse primer이다.
primer의 sq 확인 가능하다.
L.P:
atgcatcattggggaaatgtR.P:
gcaggttggtgaaagttggtAmplicon size는 product size에서 확인 가능하다. (220개)
Nucleotide position은 301번부터 복붙했는데 사이트 내에서 301번이 1번으로 바뀜
-> 앞에 300개의 n.t가 생략됨
따라서 start에 +300을 해준 값으로 생각하면 된다.
EX)
left primer start : 1639
Nucleotide position: (1939~1959)가 된다.
-> len이 20개라서
각 primer의 Tm값도 확인할 수 있다.
다음으로 qPCR에서 사용하는 TaqMan probe를 확인하고자 한다.
primer3에서 켜줬던 pick hydridization probe가 probe를 디자인 해주기 위함이었다.
probe의 sq은
caactacgcagcaactccaaNucleotide position도 다른 primer와 동일하게 +300을 해준 값으로 계산한다.
EX)
Nucleotide position: (2064~2083)임을 알 수 있다.
'전공 활동 > 실험이론' 카테고리의 다른 글
| MTT assay (0) | 2024.11.24 |
|---|---|
| 세포 배양 시 passage 측정 (0) | 2024.11.15 |
| [정제공정] IMAC (1) | 2024.11.12 |
| sprouting assay 실험 기록 (0) | 2024.11.10 |
| real-time PCR primer 디자인 (2) (2) | 2024.03.20 |



